Тракторист В,С,D,E | Учебный центр №266
Основные рабочие категории по этой профессии в удостоверении тракториста-машиниста (УТМ)
категория «B» — гусеничные и колесные машины с двигателем мощностью до 25,7 кВт
категория «C» — колесные машины с двигателем мощностью от 25,7 до 110,3 кВт
категория «D» — колесные машины с двигателем мощностью свыше 110,3 кВт
категория «E» — гусеничные машины с двигателем мощностью свыше 25,7 кВт
О программе профессиональной подготовки
Код профессии: 19203
Квалификационные разряды: 2-8
Цель обучения: дать слушателям знания, умения и навыки в формировании компетенции для выполнения трудовой деятельности трактористом.
Срок обучения: 2 месяца.
Объем программы: 55 часов, из них:
— теоретическое обучение – 40 часов;
— обучение на производстве – 8 часов.
Язык обучения: русский
Возраст: с 18 лет
Ограничения по уровню образования: отсутствуют
Место обучения: вы можете пройти обучение и получить официальный документ о квалификации независимо от места вашего проживания.
Форма обучения: заочная (очно-заочная) с применением электронного обучения и дистанционных образовательных технологий.
Заочная часть: теоретическую часть слушатель изучает самостоятельно на сайте https://экзон.рф/ в соответствующем разделе; экзамен проводится очно в «Центре №266».
| + | = | ||||
|
Программа состоит из 5 разделов Пройдите разделы (5) в предложенном порядке. |
Практическое обучение на полигоне Выполните практическую квалификационную работу. |
Квалификационный зачет по теоритической и практической части |
Свидетельство о профессии рабочего Получите документ о квалификации и приступайте к трудовой деятельности. |
* Очно-заочная форма обучения — для граждан проживающих на территории г. Тамбова и Тамбовской области.
Тракторист 2-го разряда — тракторы с двигателем мощностью до до 35 л.с.
Тракторист 3-го разряда — тракторы с двигателем мощностью свыше 35 до 60 л.с.
Тракторист 4-го разряда — тракторы с двигателем мощностью свыше 60 до 100 л. с.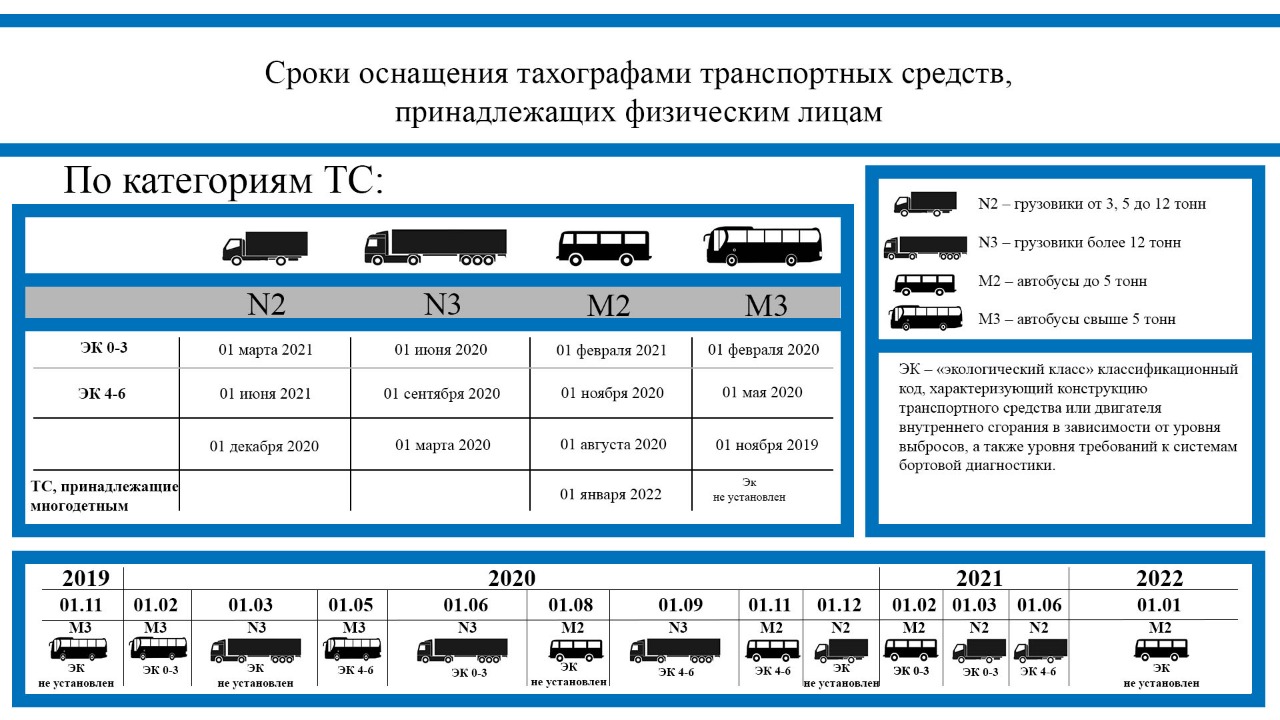
Тракторист 5-го разряда — тракторы с двигателем мощностью свыше 100 до 200 л. с.
Тракторист 6-го разряда — тракторы с двигателем мощностью двигателя свыше 200 до 380 л. с.
Тракторист 7-го разряда — тракторы с двигателем мощностью свыше 380 до 500 л. с.
Тракторист 8-го разряда — тракторы с двигателем мощностью 500 л.с. и более.
ДОКУМЕНТ ПОДТВЕРЖДАЮЩИЙ ВАШУ КВАЛИФИКАЦИЮ
После окончания образовательной программы слушатель получает документ о квалификации — свидетельство о профессии рабочего.
 № 273-ФЗ)
№ 273-ФЗ)Документ о квалификации дает право обратиться в инспекцию гостехнадзора за предоставлением государственной услуги по приему экзаменов на право управления самоходными машинами и выдачи удостоверения тракториста-машиниста (тракториста).
Классификация включенных и исключенных экзонов в случае пропуска экзона с использованием модификаций гистонов экспрессия генов (Tilgner et al., 2012). В некоторых случаях сплайсинг РНК может создавать ряд уникальных белков путем организации экзонов одной и той же пре-мРНК разными способами (Black, 2003). Это явление известно как альтернативный сплайсинг. Среди многочисленных способов альтернативного сплайсинга наиболее распространенным является пропуск экзона, при котором конкретный экзон может быть включен в мРНК при одних условиях и исключен из мРНК при других (Black, 2003).
Было продемонстрировано, что ~95% генов человека подвергаются альтернативному сплайсингу (Wang et al., 2008a). Множественные варианты транскриптов альтернативного сплайсинга из одного гена часто имеют разные биологические функции. Однако наши знания о регуляторном механизме альтернативного сплайсинга далеки от удовлетворительных.
Однако наши знания о регуляторном механизме альтернативного сплайсинга далеки от удовлетворительных.
В последние десятилетия был проведен ряд исследований с целью выявления механизмов альтернативного сплайсинга и показано, что альтернативный сплайсинг регулируется не только на уровне генома, но и на уровне эпигенома (Fox-Walsh и Fu , 2010). На уровне генома существуют экзонные и интронные энхансеры сплайсинга (ESE и ISE) и сайленсеры (ESS и ISS), представляющие собой мотивы последовательностей, которые могут распознаваться и связываться белками (Wang and Burge, 2008; Barash et al., 2010). ). Хотя информация на уровне генома может объяснить некоторые события сплайсинга, ее недостаточно для специфичного для типа клеток и специфичного для типа стадии сплайсинга РНК (Wang et al., 2008a).
Недавние исследования показали, что модификации гистонов на уровне эпигенома также участвуют в медикаментозном сплайсинге РНК. Например, Луко и др. продемонстрировали, что альтернативный сплайсинг гена FGFR2 (рецептор 2 фактора роста фибробластов) регулируется с помощью h4K36me3 (Luco et al. , 2010). Чжоу и др. обнаружили, что событие включения экзона гена человеческого фибронектина (FN1) лечится h4K9me2 и h4K27me3 (Zhou et al., 2014). Шиндо и др. обнаружили, что комбинаторный эффект модификаций гистонов также способствует альтернативным паттернам сплайсинга среди различных клеточных линий (Shindo et al., 2013). Эти результаты намекают нам, что обнаружение кода сплайсинга из модификаций гистонов даст новое понимание механизмов регуляции сплайсинга РНК.
, 2010). Чжоу и др. обнаружили, что событие включения экзона гена человеческого фибронектина (FN1) лечится h4K9me2 и h4K27me3 (Zhou et al., 2014). Шиндо и др. обнаружили, что комбинаторный эффект модификаций гистонов также способствует альтернативным паттернам сплайсинга среди различных клеточных линий (Shindo et al., 2013). Эти результаты намекают нам, что обнаружение кода сплайсинга из модификаций гистонов даст новое понимание механизмов регуляции сплайсинга РНК.
Соответственно, было предложено несколько вычислительных методов для классификации включенных и исключенных экзонов в случае пропуска экзона на основе модификаций гистонов. В 2012 году Энрот и соавт. разработали модель на основе правил и получили точность 72% (Enroth et al., 2012). Позже Чен и др. предложил метод, основанный на функции квадратичного дискриминанта (QD), и получил точность 68,5% (Chen et al., 2014). Совсем недавно, объединив особенности геномных последовательностей и модификаций гистонов, Xu et al. предложили подход глубокого обучения для прогнозирования паттернов сплайсинга (Xu et al. , 2017). Эти работы способствуют прогрессу в исследованиях по выявлению регуляторных механизмов сплайсинга РНК. Однако эффективность этих методов остается неудовлетворительной.
, 2017). Эти работы способствуют прогрессу в исследованиях по выявлению регуляторных механизмов сплайсинга РНК. Однако эффективность этих методов остается неудовлетворительной.
В текущем исследовании мы предложили новый метод классификации включенных и исключенных экзонов в случае пропуска экзона. Техника выбора признаков максимального релевантного расстояния (MRMD) использовалась для отсеивания оптимальных признаков модификации гистонов. Используя информацию о модификации гистонов, был выполнен случайный лес (RF) для создания модели прогнозирования. Результаты 10-кратного теста перекрестной проверки показывают, что предложенный метод надежен.
Материалы и методы
Набор данных
Набор данных, используемый для обучения и тестирования прогностической модели, был создан Enroth et al. (Энрот и др., 2012). Согласно данным об экспрессии генов CD4 + Т-клетки, Enroth et al. получили 13 374 «включенных» и 11 587 «исключенных» экзонов из события пропуска экзонов генома человека (Enroth et al. , 2012). Все эти экзоны имеют длину 50 п.н. с фланкирующими интронами длиной более 360 п.н., и ни один из них не перекрывается друг с другом. Энрот и др. далее нанесли на карту 20 видов ацетилирования гистонов (Barski et al., 2007) и 18 видов метилирования гистонов (Wang et al., 2008b) для этих экзонов и их ближайших 180 п.н. фланкирующих интронных областей. Сделав это, они получили сигналы модификации гистонов и представили их бинарными атрибутами, а именно наличием (обозначено «1») и отсутствием (обозначено «0») в трех областях (предшествующих, расположенных и следующих за экзонами). После удаления экзонов без модификации ацетилирования или метилирования гистонов был получен эталонный набор данных, содержащий 12,69Было получено 2 «включенных» экзона и 11 165 «исключенных» экзонов с информацией об ацетилировании и метилировании гистонов.
, 2012). Все эти экзоны имеют длину 50 п.н. с фланкирующими интронами длиной более 360 п.н., и ни один из них не перекрывается друг с другом. Энрот и др. далее нанесли на карту 20 видов ацетилирования гистонов (Barski et al., 2007) и 18 видов метилирования гистонов (Wang et al., 2008b) для этих экзонов и их ближайших 180 п.н. фланкирующих интронных областей. Сделав это, они получили сигналы модификации гистонов и представили их бинарными атрибутами, а именно наличием (обозначено «1») и отсутствием (обозначено «0») в трех областях (предшествующих, расположенных и следующих за экзонами). После удаления экзонов без модификации ацетилирования или метилирования гистонов был получен эталонный набор данных, содержащий 12,69Было получено 2 «включенных» экзона и 11 165 «исключенных» экзонов с информацией об ацетилировании и метилировании гистонов.
Образец состава
Используя бинарные атрибуты 20 видов ацетилирования гистонов и 18 видов метилирования гистонов (дополнительная таблица S1), образцы в наборе данных могут быть представлены 114-мерным вектором, заданным как
R= [ Φ1, Φ2, Φ3,⋯ Φi,⋯, Φ114]T (1), где T — оператор транспонирования. Значения компоненты вектора Φ i может быть «1» (указывает на наличие модификации гистонов) или «0» (указывает на отсутствие модификации гистонов). Φ 1 , Φ 2 , и Φ 3 указывают на присутствие или отсутствие h4K27me3 в предшествующих и последующих экзонах соответственно; Φ 4 , Φ 5 , и Φ 6 указывают на наличие или отсутствие информации для h4K4me2 и так далее. Более подробную информацию можно найти в дополнительной таблице S1. Образцы, закодированные с использованием информации о модификации гистонов, доступны по адресу https://github.com/chenweiimu/splicing.
Значения компоненты вектора Φ i может быть «1» (указывает на наличие модификации гистонов) или «0» (указывает на отсутствие модификации гистонов). Φ 1 , Φ 2 , и Φ 3 указывают на присутствие или отсутствие h4K27me3 в предшествующих и последующих экзонах соответственно; Φ 4 , Φ 5 , и Φ 6 указывают на наличие или отсутствие информации для h4K4me2 и так далее. Более подробную информацию можно найти в дополнительной таблице S1. Образцы, закодированные с использованием информации о модификации гистонов, доступны по адресу https://github.com/chenweiimu/splicing.
Выбор признаков
Если экзоны представлены вектором из 114 измерений, это может выявить следующие три неблагоприятные проблемы (Feng et al., 2013): (1) включение избыточной или нерелевантной информации; (2) что приводит к проблемам переобучения и снижению обобщающей способности модели; (3) увеличение времени вычислений. Чтобы уменьшить количество нерелевантных признаков, был предложен ряд эффективных методов выбора признаков, таких как дисперсионный анализ (Lin and Ding, 2011; Lin et al., 2015), минимальная избыточность, максимальная релевантность (Peng et al., 2005; Chen et al., 2014) и Diffusion Maps (Coifman et al., 2005).
Чтобы уменьшить количество нерелевантных признаков, был предложен ряд эффективных методов выбора признаков, таких как дисперсионный анализ (Lin and Ding, 2011; Lin et al., 2015), минимальная избыточность, максимальная релевантность (Peng et al., 2005; Chen et al., 2014) и Diffusion Maps (Coifman et al., 2005).
В этом исследовании для выбора оптимальных признаков применялся подход «Максимальная релевантность — максимальное расстояние» (MRMD), который широко используется в области биоинформатики с момента его предложения в 2016 году (Zou et al., 2016). Как указано Zou et al. (2016), основной задачей MRMD является поиск своего рода метрики ранжирования функций, которая содержит два аспекта: один — это соответствие между набором подфункций и целевым классом, а другой — избыточность набора подфункций. Более подробную информацию о MRMD можно найти в работе Zou et al., 2016 г.
Случайный лес
Случайный лес (RF) представляет собой ансамбль большого количества деревьев решений (Breiman, 2001). Каждое дерево в ансамбле обучается на подмножестве обучающих экземпляров, которые выбираются случайным образом из заданного обучающего набора. Вместо использования всех функций выбирается случайное подмножество функций, что еще больше рандомизирует дерево. Результаты прогнозирования RF основаны на ансамбле этих деревьев решений, и каждое дерево дает результат классификации. Наконец, классификатор RF выбирает результат прогнозирования, который имеет наибольшее количество голосов из результатов классификации. Благодаря своим преимуществам при работе с многомерными данными RF используется в различных областях биоинформатики (Ferrat et al., 2018; Manavalan et al., 2018; Wang et al., 2018).
Каждое дерево в ансамбле обучается на подмножестве обучающих экземпляров, которые выбираются случайным образом из заданного обучающего набора. Вместо использования всех функций выбирается случайное подмножество функций, что еще больше рандомизирует дерево. Результаты прогнозирования RF основаны на ансамбле этих деревьев решений, и каждое дерево дает результат классификации. Наконец, классификатор RF выбирает результат прогнозирования, который имеет наибольшее количество голосов из результатов классификации. Благодаря своим преимуществам при работе с многомерными данными RF используется в различных областях биоинформатики (Ferrat et al., 2018; Manavalan et al., 2018; Wang et al., 2018).
Перекрестная проверка
В статистическом прогнозировании для оценки ожидаемой вероятности успеха предиктора часто используются три метода перекрестной проверки, а именно тест независимого набора данных, тест подвыборки (или n-кратная перекрестная проверка) и тест складного ножа. . Среди трех методов перекрестной проверки тест складного ножа считается наименее произвольным и наиболее объективным (Chen et al. , 2015, 2018; Feng et al., 2018). Однако, чтобы сократить время вычислений, для оценки производительности предложенного метода был использован 10-кратный тест перекрестной проверки. Для 10-кратной перекрестной проверки набор обучающих данных случайным образом разбивается на десять обучающих подмножеств, причем девять подмножеств использовались для обучения, а оставшийся — для тестирования. Этот процесс повторялся десять раз таким образом, чтобы гарантировать, что каждый набор используется один раз для тестирования модели, которая была обучена на других девяти.
, 2015, 2018; Feng et al., 2018). Однако, чтобы сократить время вычислений, для оценки производительности предложенного метода был использован 10-кратный тест перекрестной проверки. Для 10-кратной перекрестной проверки набор обучающих данных случайным образом разбивается на десять обучающих подмножеств, причем девять подмножеств использовались для обучения, а оставшийся — для тестирования. Этот процесс повторялся десять раз таким образом, чтобы гарантировать, что каждый набор используется один раз для тестирования модели, которая была обучена на других девяти.
Оценка эффективности
Эффективность предлагаемого метода оценивалась с использованием следующих четырех показателей, а именно чувствительности ( Sn ), специфичности ( Sp ), точности ( Acc ) и коэффициента корреляции Мэтью ( MCC ), которые выражаются как (Chen et al., 2017; Lin et al., 2017; Jia et al., 2018; Zeng et al., 2018)
{Sn=TPTP+FN×100%[2 мм ]Sp=TNTN+FP×100%[2мм]Acc=TP+TNTP+FN+TN+FP×100%[2мм]MCC=(TP×TN)−(FP×FN)(TP+FN)×(TP +FP)×(TN+FN)×(TN+FP) (2), где TP, TN, FP и FN представляют истинно положительный, истинно отрицательный, ложноположительный и ложноотрицательный результат соответственно.
Результаты и обсуждение
Оценка эффективности
Путем кодирования включенных и исключенных экзонов в наборе данных с использованием модификации гистонов каждый образец был представлен 114-мерным вектором (уравнение 1), используемым в качестве входного вектора RF для построить расчетную модель. Изучив производительность модели с помощью 10-кратного теста перекрестной проверки, мы получили точность 63,49.%, что все еще далеко от нашего удовлетворения. Чтобы повысить производительность предлагаемой модели, необходимо выбрать оптимальное количество признаков для построения надежной и эффективной прогностической модели.
Поэтому мы использовали MRMD вместе со стратегией постепенного выбора функций (IFS) для создания оптимальных подмножеств функций. Мы ранжировали 114 признаков с помощью алгоритма MRMD. Затем 114 ранжированных функций были добавлены одна за другой от более низкого к более высокому рангу. Эта процедура повторялась 114 раз, и для каждого раза строилась РЧ-модель. Их производительность была исследована с помощью 5-кратного теста перекрестной проверки. Наиболее оптимальные характеристики могут быть получены, когда точность достигает своего максимума. IFS использовался для определения оптимального количества признаков. Соответствующая кривая IFS была построена на рисунке 1. Точность достигает своего максимума 790,79%, когда для кодирования образцов использовались 96 признаков с наивысшим рейтингом. Поэтому на основе этих 96 оптимальных признаков была построена вычислительная модель. В этом случае предложенная модель получила точность 72,91% при чувствительности 67,03% и специфичности 79,65% в 10-кратном тесте перекрестной проверки.
Их производительность была исследована с помощью 5-кратного теста перекрестной проверки. Наиболее оптимальные характеристики могут быть получены, когда точность достигает своего максимума. IFS использовался для определения оптимального количества признаков. Соответствующая кривая IFS была построена на рисунке 1. Точность достигает своего максимума 790,79%, когда для кодирования образцов использовались 96 признаков с наивысшим рейтингом. Поэтому на основе этих 96 оптимальных признаков была построена вычислительная модель. В этом случае предложенная модель получила точность 72,91% при чувствительности 67,03% и специфичности 79,65% в 10-кратном тесте перекрестной проверки.
Рисунок 1 . Кривая IFS для классификации «включенных» и «исключенных» экзонов в случае пропуска экзона. Пик IFS 79,79% был получен при использовании оптимальных 96 признаков для выполнения прогнозов.
Сравнительный анализ различных классификаторов
Чтобы дополнительно продемонстрировать эффективность предложенного метода классификации «включенных» и «исключенных» экзонов, мы сравнили его эффективность с эффективностью других классификаторов, таких как BayseNet, Naïve Bayes, J48 Tree и Машина опорных векторов (SVM).
Таблица 1 . Показатели производительности различных классификаторов для классификации включенных и исключенных экзонов.
Кроме того, было также проведено сравнение между текущим методом и методом в нашей предыдущей работе (Chen et al., 2014), где был предложен метод на основе функции QD для классификации «включенных» и «исключенных» экзонов. Поскольку оба метода обучаются и тестируются на основе одного и того же набора данных, мы напрямую сравнили 10-кратные результаты перекрестной проверки текущего метода с результатами, указанными в предыдущей работе (Chen et al.
Таблица 2 . Сравнение текущего метода с существующим методом классификации включенных и исключенных экзонов.
Анализ признаков
Чтобы получить общее представление об оптимальных признаках для классификации «включенных» и «исключенных» экзонов, мы сравнили их частотные распределения в обоих типах экзонов, используя тест
 Этот результат указывает на то, что основные регуляторные эпигенетические факторы события пропуска экзона локализованы в областях, окружающих экзоны.
Этот результат указывает на то, что основные регуляторные эпигенетические факторы события пропуска экзона локализованы в областях, окружающих экзоны.Таблица 3 . 96 оптимальных признаков и их склонность к включению или исключению экзона, случай a .
Недавние исследования показали, что сплайсинг РНК может регулироваться не только модификацией одного типа гистонов, но и комбинацией различных типов модификаций гистонов (Shindo et al., 2013). Чтобы определить, существует ли кооперация или конкуренция модификаций гистонов в процессе пропуска экзонов, мы рассчитали коэффициент корреляции Пирсона для 81 оптимального признака. Корреляционная матрица для «включенных» и «исключенных» экзонов была представлена на рисунках 2 и 3 соответственно. Как показано на этих рисунках, между различными видами модификаций гистонов можно наблюдать значительные положительные и отрицательные корреляции. Например, в случае «включенного» экзона h4K18ac положительно коррелирует с h4K23ac, h5K8ac и h5K12ac, тогда как h5K91ac отрицательно коррелирует с h4K91me2.
Рисунок 2 . Корреляционная матрица модификаций гистонов для случая включения экзона в случае пропуска экзона.
Рисунок 3 . Корреляционная матрица модификаций гистонов для случая исключения экзона при пропуске экзона.
Заключение
Являясь одним из ключевых процессов генной экспрессии, помимо ESE, ISE, ESS, ISS и других транс-элементов, сплайсинг РНК также регулируется эпигенетическими факторами. В этой статье мы представили новый вычислительный метод для классификации «включенных» и «исключенных» экзонов в событиях пропуска экзонов на основе модификаций гистонов. Образцы в наборе данных были закодированы с использованием информации об оптимальной модификации гистонов, полученной методом выбора признаков, а затем использованы в качестве входных данных RF. Прогностические результаты, полученные с помощью 10-кратного теста перекрестной проверки, показали, что предлагаемый подход может обеспечить более высокую производительность, чем существующие подходы.
Прогностические результаты, полученные с помощью 10-кратного теста перекрестной проверки, показали, что предлагаемый подход может обеспечить более высокую производительность, чем существующие подходы.
Чтобы обеспечить интуитивное представление о модификациях гистонов, которые вносят вклад в предсказания, мы систематически анализировали их распределения в «включенных» и «исключенных» экзонах. Неслучайное распределение модификаций гистонов (таблица 3) и их профили положительной или отрицательной корреляции (рис. 2, 3) позволяют предположить, что пропуск экзонов регулируется комбинацией различных типов модификаций гистонов. Необходимы дальнейшие экспериментальные исследования, чтобы показать, как эти модификации гистонов связаны со сплайсингом.
В будущей работе мы сделаем все возможное, чтобы разработать гораздо более умный метод классификации «включенных» и «исключенных» экзонов путем интеграции информации как с геномного, так и с эпигеномного уровней.
Вклад авторов
WC и HL задумали и разработали эксперименты. PF и HD провели эксперименты. HL и WC написали статью. Все авторы прочитали и одобрили окончательный вариант рукописи.
PF и HD провели эксперименты. HL и WC написали статью. Все авторы прочитали и одобрили окончательный вариант рукописи.
Финансирование
Работа выполнена при поддержке Национального научного фонда Китая (31771471, 61772119), Фонд естественных наук для выдающихся молодых ученых провинции Хэбэй (№ C2017209244), Программа для лучших молодых инновационных талантов высших учебных заведений провинции Хэбэй (№ BJ2014028).
Заявление о конфликте интересов
Авторы заявляют, что исследование проводилось при отсутствии каких-либо коммерческих или финансовых отношений, которые могли бы быть истолкованы как потенциальный конфликт интересов.
Дополнительный материал
Дополнительный материал к этой статье можно найти в Интернете по адресу: https://www.frontiersin.org/articles/10.3389/fgene.2018.00433/full#supplementary-material
Ссылки
Barash, Y., Calarco, J.A., Gao, W., Pan, Q., Wang, X., Shai, O., et al. (2010). Расшифровка кода сплайсинга. Природа 465, 53–59. doi: 10.1038/nature09000
Природа 465, 53–59. doi: 10.1038/nature09000
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Барски А., Куддапах С., Цуй К., Рох Т.Ю., Шоунс Д.Е., Ван З. и др. (2007). Профилирование метилирования гистонов в геноме человека с высоким разрешением. Ячейка 129, 823–837. doi: 10.1016/j.cell.2007.05.009
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Black, DL (2003). Механизмы альтернативного сплайсинга пре-мессенджерных РНК. год. Преподобный Биохим. 72, 291–336. doi: 10.1146/annurev.biochem.72.121801.161720
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Брейман, Л. (2001). Случайные леса. Маха. Учиться. 45, 5–32. doi: 10.1023/A:1010933404324
Полный текст CrossRef | Академия Google
Chen, W., Feng, P., Ding, H., Lin, H., and Chou, K.C. (2015). iRNA-Methyl: идентификация сайтов N(6)-метиладенозина с использованием псевдонуклеотидной композиции. Анал. Биохим. 490, 26–33. doi: 10.1016/j.ab.2015.08.021.
Анал. Биохим. 490, 26–33. doi: 10.1016/j.ab.2015.08.021.
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Чен В., Фэн П., Ян Х., Дин Х., Лин Х. и Чоу К. К. (2018). iRNA-3typeA: идентификация трех типов модификации аденозиновых сайтов РНК. Мол. тер. Нуклеиновые кислоты 11, 468–474. doi: 10.1016/j.omtn.2018.03.012.
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Чен В., Лин Х., Фэн П. и Ван Дж. (2014). Предсказание события пропуска экзона на основе модификаций гистонов. Междисциплинарный. науч. 6, 241–249. doi: 10.1007/s12539-013-0195-4.
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Чен В., Ян Х., Фэн П., Дин Х. и Линь Х. (2017). iDNA4mC: идентификация сайтов ДНК N4-метилцитозин на основе химических свойств нуклеотидов. Биоинформатика 33, 3518–3523. doi: 10.1093/биоинформатика/btx479.
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Койфман Р. Р., Лафон С., Ли А. Б., Маджони М., Надлер Б., Уорнер Ф. и др. (2005). Геометрические диффузии как инструмент гармонического анализа и определения структуры данных: карты диффузии. Проц. Натл. акад. науч. США 102, 7426–7431. doi: 10.1073/pnas.0500334102
Р., Лафон С., Ли А. Б., Маджони М., Надлер Б., Уорнер Ф. и др. (2005). Геометрические диффузии как инструмент гармонического анализа и определения структуры данных: карты диффузии. Проц. Натл. акад. науч. США 102, 7426–7431. doi: 10.1073/pnas.0500334102
PubMed Abstract | Полный текст перекрестной ссылки | Академия Google
Энрот, С., Борнелов, С., Ваделиус, К., и Коморовски, Дж. (2012). Комбинации модификаций гистонов маркируют уровни включения экзонов. PLoS ONE 7:e29911. doi: 10.1371/journal.pone.0029911
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Фэн П., Ян Х., Дин Х., Линь Х., Чен В. и Чоу К. К. (2018). iDNA6mA-PseKNC: идентификация сайтов N(6)-метиладенозина ДНК путем включения физико-химических свойств нуклеотидов в PseKNC. Геномика doi: 10.1016/j.ygeno.2018.01.005. [Epub перед печатью].
Полнотекстовая перекрестная ссылка | Google Scholar
Фэн П. М., Чен В., Лин Х. и Чоу К. К. (2013). iHSP-PseRAAAC: идентификация семейств белков теплового шока с использованием состава псевдосокращенного аминокислотного алфавита. Анал. Биохим. 442, 118–125. doi: 10.1016/j.ab.2013.05.024
iHSP-PseRAAAC: идентификация семейств белков теплового шока с использованием состава псевдосокращенного аминокислотного алфавита. Анал. Биохим. 442, 118–125. doi: 10.1016/j.ab.2013.05.024
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Феррат Л. А., Гудфеллоу М. и Терри Дж. Р. (2018). Классификация динамических переходов в многомерных моделях нейронной массы: метод случайного леса. Вычисление PLoS. биол. 14:e1006009. doi: 10.1371/journal.pcbi.1006009
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Фокс-Уолш, К., и Фу, X. Д. (2010). Хроматин: последний рубеж в регуляции сплайсинга? Дев. Ячейка 18, 336–338. doi: 10.1016/j.devcel.2010.03.002
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Фрэнк Э., Холл М., Тригг Л., Холмс Г. и Виттен И. Х. (2004). Интеллектуальный анализ данных в биоинформатике с использованием Weka. Биоинформатика 20, 2479–2481. doi: 10.1093/bioinformatics/bth361
doi: 10.1093/bioinformatics/bth361
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Цзя, К., Цзо, Ю. и Цзоу, К. (2018). O-GlcNAcPRED-II: интегрированный алгоритм классификации для определения сайтов O-GlcNAcylation на основе нечеткой недостаточной выборки и метода передискретизации PCA K-средних. Биоинформатика 34, 2029–2036. doi: 10.1093/bioinformatics/bty039
PubMed Abstract | Полный текст перекрестной ссылки | Академия Google
Лин Х. и Дин Х. (2011). Прогнозирование ионных каналов и их типов по дипептидному типу псевдоаминокислотного состава. Ж. Теор. биол. 269, 64–69. doi: 10.1016/j.jtbi.2010.10.019
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Лин Х., Лян З. Ю., Тан Х. и Чен В. (2017). Идентификация промоторов сигма70 с новым псевдонуклеотидным составом. IEEE/ACM Trans. вычисл. биол. Биоинформ. doi: 10.1109/TCBB.2017.2666141. [Epub перед печатью].
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Лин Х. , Лю В. Х., Хе Дж., Лю Х. Х., Дин Х. и Чен В. (2015). Прогнозирование раковых лектинов с помощью оптимальных дипептидов g-gap. Науч. Респ. 5:16964. doi: 10.1038/srep16964
, Лю В. Х., Хе Дж., Лю Х. Х., Дин Х. и Чен В. (2015). Прогнозирование раковых лектинов с помощью оптимальных дипептидов g-gap. Науч. Респ. 5:16964. doi: 10.1038/srep16964
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Луко Р. Ф., Пан К., Томинага К., Бленкоу Б. Дж., Перейра-Смит О. М. и Мистели Т. (2010). Регуляция альтернативного сплайсинга модификациями гистонов. Наука 327, 996–1000. doi: 10.1126/science.1184208
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Манавалан Б., Шин Т. Х., Ким М. О. и Ли Г. (2018). AIPpred: предсказание противовоспалительных пептидов на основе последовательности с использованием случайного леса. Перед. Фармакол. 9:276. doi: 10.3389/fphar.2018.00276
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Пэн Х., Лонг Ф. и Дин К. (2005). Выбор признаков на основе взаимной информации: критерии максимальной зависимости, максимальной релевантности и минимальной избыточности. IEEE Trans. Анальный узор. Мах. Интел. 27, 1226–1238. doi: 10.1109/TPAMI.2005.159
IEEE Trans. Анальный узор. Мах. Интел. 27, 1226–1238. doi: 10.1109/TPAMI.2005.159
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Синдо Ю., Нодзаки Т., Сайто Р. и Томита М. (2013). Компьютерный анализ ассоциаций между альтернативным сплайсингом и модификациями гистонов. ФЭБС Письмо. 587, 516–521. doi: 10.1016/j.febslet.2013.01.032
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Tilgner, H., Knowles, D.G., Johnson, R., Davis, C.A., Chakrabortty, S., Djebali, S., et al. (2012). Глубокое секвенирование фракций субклеточной РНК показывает, что сплайсинг является преимущественно ко-транскрипционным в геноме человека, но неэффективен для lncRNAs. Рез. генома. 22, 1616–1625. doi: 10.1101/gr.134445.111
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ван Э. Т., Сандберг Р., Луо С., Хребтукова И., Чжан Л., Майр К. и др. (2008а). Альтернативная регуляция изоформ в транскриптомах тканей человека. Природа 456, 470–476. doi: 10.1038/nature07509
Природа 456, 470–476. doi: 10.1038/nature07509
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ван, X., Лин, П., и Хо, JWK (2018). Открытие грамматики мотивов ДНК, специфичных для клеточного типа, в цис-регуляторных элементах с использованием случайного леса. BMC Genomics 19 (Приложение 1): 929. doi: 10.1186/s12864-017-4340-z
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Wang, Z., and Burge, CB (2008). Регулирование сплайсинга: от перечня элементов регулирования до интегрированного кода сплайсинга. РНК 14, 802–813. doi: 10.1261/rna.876308
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Wang, Z., Zang, C., Rosenfeld, J. A., Schones, D. E., Barski, A., Cuddapah, S., et al. (2008б). Комбинаторные закономерности ацетилирования и метилирования гистонов в геноме человека. Нац. Жене. 40, 897–903. doi: 10.1038/ng.154
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Сюй Ю. , Ван Ю., Луо Дж., Чжао В. и Чжоу Х. (2017). Глубокое изучение сплайсингового (эпи)генетического кода выявило новый механизм-кандидат, связывающий модификации гистонов с решением судьбы ЭСК. Рез. нуклеиновых кислот. 45, 12100–12112. doi: 10.1093/nar/gkx870
, Ван Ю., Луо Дж., Чжао В. и Чжоу Х. (2017). Глубокое изучение сплайсингового (эпи)генетического кода выявило новый механизм-кандидат, связывающий модификации гистонов с решением судьбы ЭСК. Рез. нуклеиновых кислот. 45, 12100–12112. doi: 10.1093/nar/gkx870
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Цзэн X., Лю Л., Лу Л. и Цзоу К. (2018). Прогнозирование микроРНК, потенциально связанных с заболеванием, с использованием метода структурных возмущений. Биоинформатика 34, 2425–2432. doi: 10.1093/bioinformatics/bty112
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Чжоу, Х. Л., Луо, Г., Уайз, Дж. А., и Лу, Х. (2014). Регуляция альтернативного сплайсинга локальными модификациями гистонов: потенциальная роль механизмов, управляемых РНК. Рез. нуклеиновых кислот. 42, 701–713. doi: 10.1093/nar/gkt875
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Цзоу, К. , Цзэн, Дж. К., Цао, Л. Дж., и Цзэн, X. X. (2016). Новая метрика ранжирования с применением масштабируемой классификации визуальных и биоинформатических данных. Нейрокомпьютинг 173, 346–354. doi: 10.1016/j.neucom.2014.12.123
, Цзэн, Дж. К., Цао, Л. Дж., и Цзэн, X. X. (2016). Новая метрика ранжирования с применением масштабируемой классификации визуальных и биоинформатических данных. Нейрокомпьютинг 173, 346–354. doi: 10.1016/j.neucom.2014.12.123
CrossRef Полный текст | Google Scholar
Эукариотические факторы высвобождения — wikidoc
Эукариотический фактор терминации трансляции 1 (eRF1), также известный как TB3-1 , представляет собой белок, который у человека кодируется геном ETF1 . [1] [2] [3]
У эукариот это единственный фактор высвобождения (eRF), который распознает все три стоп-кодона. Общий процесс терминации подобен у прокариот, но у последних существуют 3 отдельных фактора высвобождения, RF1, RF2 и RF3. [4]
Содержание
- 1 Функция
- 2 Каталожные номера
- 3 Дополнительная литература
- 4 Внешние ссылки
Функция
Об окончании биосинтеза белка и высвобождении формирующейся полипептидной цепи сигнализирует наличие внутрирамочного стоп-кодона в аминоацильном участке рибосомы. Процесс терминации трансляции универсален и опосредуется факторами высвобождения белка (RF) и GTP. RF класса 1 распознает стоп-кодон и способствует гидролизу сложноэфирной связи, связывающей полипептидную цепь с пептидильным участком тРНК, реакции, катализируемой пептидилтрансферазным центром рибосомы. РФ класса 2, которые не являются кодон-специфичными и не распознают кодоны, стимулируют активность РФ класса 1 и придают GTP-зависимость от процесса. У прокариот оба RF класса 1, RF1 и RF2, распознают UAA; однако UAG и UGA декодируются именно RF1 и RF2 соответственно. У эукариот eRF1 или ETF1, функциональный аналог RF1 и RF2, функционирует как всемогущий RF, декодируя все 3 стоп-кодона. [1] [3]
Процесс терминации трансляции универсален и опосредуется факторами высвобождения белка (RF) и GTP. RF класса 1 распознает стоп-кодон и способствует гидролизу сложноэфирной связи, связывающей полипептидную цепь с пептидильным участком тРНК, реакции, катализируемой пептидилтрансферазным центром рибосомы. РФ класса 2, которые не являются кодон-специфичными и не распознают кодоны, стимулируют активность РФ класса 1 и придают GTP-зависимость от процесса. У прокариот оба RF класса 1, RF1 и RF2, распознают UAA; однако UAG и UGA декодируются именно RF1 и RF2 соответственно. У эукариот eRF1 или ETF1, функциональный аналог RF1 и RF2, функционирует как всемогущий RF, декодируя все 3 стоп-кодона. [1] [3]
Ссылки
- ↑ 1.0 1.1 «Ген Entrez: эукариотический фактор терминации трансляции 1».
- ↑ Гренетт Х. Е., Эйперс П. Г., Кидд В. Дж., Бунелис П., Фуллер Г. М. (январь 1992 г.). «Хромосомная локализация кДНК человека, содержащая домен связывания DIDS и демонстрирующая высокую гомологию с всемогущим супрессором 45 дрожжей».
 Сомат. Ячейка Мол. Гене . 18 (1): 97–102. дои: 10.1007/BF01233452. PMID 1546371.
Сомат. Ячейка Мол. Гене . 18 (1): 97–102. дои: 10.1007/BF01233452. PMID 1546371. - ↑ 3.0 3.1 Фролова Л., Ле Гофф Х., Расмуссен Х.Х., Чеперегин С., Другеон Г., Кресс М., Арман И., Хенни А.Л., Селис Дж.Е., Филипп М. (декабрь 1994 г.). «Высококонсервативное семейство эукариотических белков, обладающее свойствами фактора высвобождения полипептидной цепи». Природа . 372 (6507): 701–3. дои: 10.1038/372701a0. PMID 7990965.
- ↑ Киселев Л., Эренберг М., Фролова Л. (январь 2003 г.). «Остановка трансляции: взаимодействие мРНК, рРНК и факторов высвобождения?». ЕМВО J . 22 (2): 175–82. doi: 10.1093/emboj/cdg017. PMC 140092. PMID 12514123.
Дополнительная литература
- Кашима И., Ямасита А., Идзуми Н., Катаока Н., Моришита Р., Хосино С., Оно М., Дрейфус Г., Оно С. (2006). «Связывание нового комплекса SMG-1-Upf1-eRF1-eRF3 (SURF) с комплексом соединения экзонов запускает фосфорилирование Upf1 и нонсенс-опосредованный распад мРНК».
 Гены Дев . 20 (3): 355–67. дои: 10.1101/gad.1389006. PMC 1361706. PMID 16452507.
Гены Дев . 20 (3): 355–67. дои: 10.1101/gad.1389006. PMC 1361706. PMID 16452507. - Шаватт Л., Сеит-Неби А., Дубовая В., Фавр А. (2002). «Инвариантный уридин стоп-кодонов связывается с консервативной петлей NIKSR человеческого eRF1 в рибосоме». EMBO J . 21 (19): 5302–11. дои: 10.1093/emboj/cdf484. PMC 129024. PMID 12356746.
- Janzen DM, Geballe AP (2004). «Влияние истощения эукариотического фактора высвобождения на терминацию трансляции в клеточных линиях человека». Рез. нуклеиновых кислот . 32 (15): 4491–502. дои: 10.1093/нар/гх791. PMC 516063. PMID 15326224.
- Руал Дж.Ф., Венкатесан К., Хао Т., Хирозане-Кишикава Т., Дрикот А., Ли Н., Берриз Г.Ф., Гиббонс Ф.Д., Дрезе М., Айиви-Гедехуссу Н., Клитгорд Н., Саймон С., Боксем М., Мильштейн С., Розенберг Дж. , Гольдберг Д.С., Чжан Л.В., Вонг С.Л., Франклин Г., Ли С., Албала Д.С., Лим Дж., Фроутон С., Лламосас Э., Чевик С.
 , Бекс С., Ламеш П., Сикорский Р.С., Ванденхаут Дж., Зогби Х.И., Смоляр А., Босак S, Sequerra R, Doucette-Stamm L, Cusick ME, Hill DE, Roth FP, Vidal M (2005). «На пути к карте в масштабе протеома сети межбелковых взаимодействий человека». Природа . 437 (7062): 1173–8. дои: 10.1038/природа04209. PMID 16189514.
, Бекс С., Ламеш П., Сикорский Р.С., Ванденхаут Дж., Зогби Х.И., Смоляр А., Босак S, Sequerra R, Doucette-Stamm L, Cusick ME, Hill DE, Roth FP, Vidal M (2005). «На пути к карте в масштабе протеома сети межбелковых взаимодействий человека». Природа . 437 (7062): 1173–8. дои: 10.1038/природа04209. PMID 16189514. - Иванова Е.В., Алкалаева Е.З., Бирсдалл Б., Колосов П.М., Полшаков В.И., Киселев Л.Л. (2008). «[Интерфейс взаимодействия среднего домена фактора терминации трансляции человека eRF1 с эукариотическими рибосомами]». Мол. биол. (Москва) . 42 (6): 1056–66. PMID 19140327.
- Гаурылюк В., Завьялов А., Киселев Л., Эренберг М. (2006). «Фактор высвобождения класса 1 eRF1 способствует связыванию GTP с помощью фактора высвобождения класса 2 eRF3». Биохимия . 88 (7): 747–57. doi: 10.1016/j.biochi.2006.06.001. PMID 16797113.
- Инге-Вечтомов С., Журавлева Г., Филипп М.
 (2003). «История эукариотических факторов высвобождения (eRF)». биол. Сотовый . 95 (3–4): 195–209. doi: 10.1016/S0248-4900(03)00035-2. PMID 12867083.
(2003). «История эукариотических факторов высвобождения (eRF)». биол. Сотовый . 95 (3–4): 195–209. doi: 10.1016/S0248-4900(03)00035-2. PMID 12867083. - Андер М., Аквист Дж. (2009 г.). «Влияет ли метилирование глутамина на внутреннюю конформацию универсально консервативного мотива GGQ в факторах высвобождения рибосом?». Биохимия . 48 (15): 3483–9. дои: 10.1021/bi7r. PMID 19265422.
- Кобаяши Ю., Чжуан Дж., Пельц С., Догерти Дж. (2010). «Идентификация клеточного фактора, который модулирует запрограммированный ВИЧ-1 сдвиг рамки считывания рибосом». Дж. Биол. Химия . 285 (26): 19776–84. doi: 10.1074/jbc.M109.085621. PMC 2888388. PMID 20418372.
- Сова М.Е., Беннетт Э.Дж., Гиги С.П., Харпер Дж.В. (2009). «Определение ландшафта взаимодействия деубиквитинирующих ферментов человека». Сотовый . 138 (2): 389–403. doi:10.1016/j.cell.2009.04.042. PMC 2716422.
 PMID 19615732.
PMID 19615732. - Илегемс Э., Пик Х.М., Фогель Х. (2004). «Подавление eRF1 с помощью РНК-интерференции увеличивает эффективность подавления неправильно ацилированной тРНК в клетках человека». Белок Eng. Дес. Сел . 17 (12): 821–7. doi: 10.1093/белок/gzh096. PMID 15716307.
- Колосов П., Фролова Л., Сеит-Неби А., Дубовая В., Кононенко А., Опарина Н., Юстесен Дж., Ефимов А., Киселев Л. (2005). «Инвариантные аминокислоты, необходимые для декодирования функции фактора высвобождения полипептида eRF1». Рез. нуклеиновых кислот . 33 (19): 6418–25. дои: 10.1093/нар/gki927. PMC 1283522. PMID 16282590.
- Андерсен Дж. С., Лам Ю. В., Леунг А. К., Онг С. Е., Лион К. Э., Ламонд А. И., Манн М. (2005). «Динамика ядрышкового протеома». Природа . 433 (7021): 77–83. дои: 10.1038/природа03207. PMID 15635413.
- Фигаро С., Скрима Н., Букингем Р. Х., Эрге-Хамар В.
 (2008 г.). «Белок HemK2, кодируемый на хромосоме 21 человека, метилирует фактор терминации трансляции eRF1». Письмо ФЭБС . 582 (16): 2352–6. doi:10.1016/j.febslet.2008.05.045. PMID 18539146.
(2008 г.). «Белок HemK2, кодируемый на хромосоме 21 человека, метилирует фактор терминации трансляции eRF1». Письмо ФЭБС . 582 (16): 2352–6. doi:10.1016/j.febslet.2008.05.045. PMID 18539146. - Шаватт Л., Фролова Л., Лаугаа П., Киселев Л., Фавр А. (2003). «Стоп-кодоны и UGG способствуют эффективному связыванию фактора высвобождения полипептида eRF1 с сайтом A рибосомы». Дж. Мол. Биол . 331 (4): 745–58. doi: 10.1016/S0022-2836(03)00813-1. PMID 12909007.
- Бонсак М.Т., Регенер К., Шваппах Б., Саффрих Р., Параскева Э., Хартманн Э., Гёрлих Д. (2002). «Exp5 экспортирует eEF1A через тРНК из ядер и взаимодействует с другими путями транспорта, ограничивая трансляцию цитоплазмой». ЕМВО J . 21 (22): 6205–15. doi: 10.1093/emboj/cdf613. PMC 137205. PMID 12426392.
- Gevaert K, Goethals M, Martens L, Van Damme J, Staes A, Thomas GR, Vandekerckhove J (2003). «Изучение протеомов и анализ процессинга белков с помощью масс-спектрометрической идентификации отсортированных N-концевых пептидов».
 Нац. Биотехнолог . 21 (5): 566–9. дои: 10.1038/nbt810. PMID 12665801.
Нац. Биотехнолог . 21 (5): 566–9. дои: 10.1038/nbt810. PMID 12665801. - Фунакоши Ю., Дои Ю., Хосода Н., Учида Н., Осава М., Шимада И., Цудзимото М., Судзуки Т., Катада Т., Хосино С. (2007). «Механизм деаденилирования мРНК: свидетельство молекулярного взаимодействия между фактором терминации трансляции eRF3 и деаденилазами мРНК». Гены Дев . 21 (23): 3135–48. doi:10.1101/gad.1597707. PMC 2081979. PMID 18056425.
- Иванова Е.В., Колосов П.М., Бердсолл Б., Келли Г., Пасторе А., Киселев Л.Л., Полшаков В.И. (2007). «Эукариотический фактор терминации трансляции класса 1 eRF1: ЯМР-структура среднего домена, участвующего в запуске рибосомозависимого гидролиза пептидил-тРНК». Журнал ФЭБС . 274 (16): 4223–37. doi:10.1111/j.1742-4658.2007.05949.x. PMID 17651434.
- Манцызов А.Б., Иванова Е.В., Бердсолл Б., Алкалаева Е.З., Крючкова П.Н., Келли Г., Фролова Л.Ю., Полшаков В.И.


